- Laboratório
- Medicina laboratorial
- Software de gestão de dados
- PhenoSystems SA

- Produtos
- Catálogos
Software para sequenciamento de NGS GensearchNGSde análisede gestão de dadosde diagnóstico
Guardar nos favoritos
Comparar
fo_shop_gate_exact_title
Características
- Função
- de análise, de gestão de dados, de diagnóstico
- Aplicações
- para sequenciamento de NGS, de laboratório
Descrição
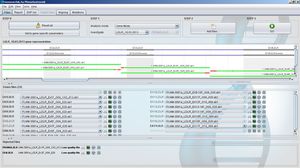
GensearchNGS é uma solução de software integrada para a análise de dados de DNA-Seq de equipamentos NGS normalmente utilizados, tais como Roche/454, Illumina, Ion Torrent e Proton. Foi concebido e desenvolvido em estreita colaboração com os principais grupos de diagnóstico credenciados na Europa e com o apoio do projecto NMDchip FP7.
Integra todas as etapas desde a importação e filtragem de leituras sequenciais, divisão de códigos de barras, alinhamento, detecção de variantes e anotação até ao relatório final das variantes detectadas. As variantes podem ser anotadas com várias fontes de dados públicas e privadas (Ensembl, Alamut, Genome Trax,...) Elas podem ser filtradas por combinações lógicas entre amostras para estudos familiares.
Características principais:
Software amigável para ressequenciar projetos NGS em uma configuração de diagnóstico, rodando em sistemas Windows, Linux e MacOS.
Conecta*) a ferramentas de interpretação de variantes e bancos de dados, tais como: Ensembl, dbSNP, Alamut, Clinvar, Wiki Pathways,Gene Ontology*).
Filtrar variantes entre amostras (por exemplo, variantes comuns a várias amostras ou excluir variantes de uma amostra de outra).
Sistema de banco de dados centralizado no paciente para gerenciar dados de seqüência e variantes validadas.
Personalização possível para exportar dados para o sistema de gestão do seu laboratório.
Possibilidade de publicar as suas variantes no Clinvar*).
Desenvolvido em conjunto com laboratórios acreditados.
Para projetos maiores (WES/WGS): possibilidade de usar vários PCs em paralelo.
Tecnologia Plugin: alinhamento com nossos algoritmos proprietários ou públicos, tais como Bowtie, BWA ou Stampy.
Anotação e filtragem com descritores e códigos HPO, OMIM e Orphanet, úteis na pesca de mutações em exomas inteiros ou genomas inteiros.
---
* Os preços não incluem impostos, transporte, taxas alfandegárias, nem custos adicionais associados às opções de instalação e de ativação do serviço. Os preços são meramente indicativos e podem variar em função dos países, do custo das matérias-primas e das taxas de câmbio.