- Laboratório
- Biologia molecular
- Módulo de software de análise
- JSI medical systems
Módulo de software para sequenciamento de NGS SEQNEXTde análisede capturade mapeamento
Guardar nos favoritos
Comparar
fo_shop_gate_exact_title
Características
- Função
- de análise, de captura, de mapeamento
- Aplicações
- de laboratório, para sequenciamento de NGS
Descrição
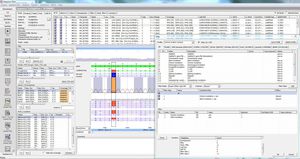
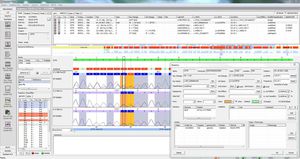
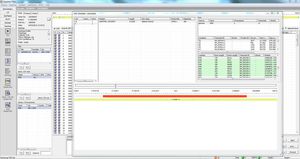
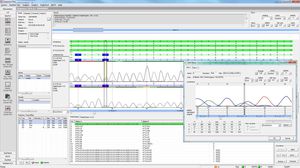
SEQNEXT é uma aplicação poderosa e de fácil utilização para cartografia, alinhamento e detecção de variantes de dados de sequenciação da Próxima Geração. A SEQNEXT pode analisar dados de todas as plataformas e kits de sequenciação comuns (baseados em PCR, enriquecimento, captura de sequências). A visualização de todas as variantes detectadas, como eliminações, inserções e indels de qualquer comprimento, SNPs, regiões repetidas, bem como CNVs e fusões de genes, é clara e intuitiva.
A SEQNEXT oferece acesso a bases de dados públicas SNP como dbSNP, 1000 Genomes, ClinVar, ExAC e gnomAD para classificação e filtragem. Todos os dados de resultados podem ser trocados com a nossa base de dados de variantes para Experiência e Conhecimento Partilhados - varSEAK, transferidos para sistemas LIM internos de laboratório e/ou emitidos como relatórios personalizados de pacientes.
NOVO Melhor análise de dados de sequenciamento de vírus, por exemplo, SARS-CoV-2
Trioanálise e poolanalise melhorada
Sequência Ontologia Anotações na tabela de variantes
Opção de salto para varSEAK e todos os DBs externos disponíveis como dbSNP, ClinVar, UK10K, ESP, gnomAD e ExAC
Análise de Sequenciação de todo o Exome (WES)
Compatível com dados de todas as plataformas e kits de sequenciação da Próxima Geração comuns
Fácil criação de regiões-alvo individuais (ROIs) através da importação de ficheiros de cama ou de manifestação com base em hg19 e/ou hg38
Importação de listas de genes para filtragem e análise de dados WES
Análise de fastq-, fastq.gz- ou bam-files
Normas eficientes para cartografia, alinhamento, qualidade e chamada de variantes (algoritmos utilizados: BWA e Smith-Waterman, adaptados pela JSI)
Configurações personalizadas para análise e chamada de variantes (≥ 0,1 %)
Alta sensibilidade e especificidade para a detecção de deleções, inserções e indels de qualquer comprimento, SNPs, regiões repetidas, bem como CNVs e fusões de genes
---
Catálogos
SEQUENCE PILOT
2 Páginas
Pesquisas relacionadas
- Software de análise
- Software de criação de laudos
- Software automático
- Módulo de software
- Software de importação
- Software servidor
- Módulo de software de análise
- Software para biologia molecular
- Módulo de software de laboratório
- Módulo de software de captura
- Módulo de software de controle
- Software NGS
- Módulo de software de mapeamento
* Os preços não incluem impostos, transporte, taxas alfandegárias, nem custos adicionais associados às opções de instalação e de ativação do serviço. Os preços são meramente indicativos e podem variar em função dos países, do custo das matérias-primas e das taxas de câmbio.